
Fusões no recetor do fator de crescimento de fibroblastos 2 (FGFR2) no colangiocarcinoma intra-hepático (iCCA)
Alterações genómicas nos recetores do fator de crescimento de fibroblastos (FGFRs)
- Os FGFRs são uma família de recetores da tirosina quinase.1,2
A via de sinalização do FGFR tem um papel central em vários processos celulares, incluindo a proliferação, a migração e a sobrevivência celular1,2
- As alterações genéticas do FGFR foram identificadas como promotores da tumorigénese em várias neoplasias, incluindo o iCCA, o carcinoma urotelial, as neoplasias mieloides/linfoides e outros tumores malignos1,3,4
- Foram observadas amplificações, mutações e fusões em todos os subtipos de FGFR (FGFR1–4).5
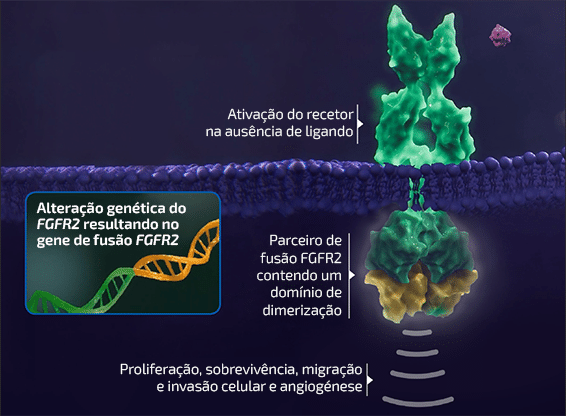
No iCCA têm sido frequentemente identificados rearranjos cromossómicos que envolvem o FGFR2,
resultando na criação de proteínas de fusão oncogénicas6
- As fusões de genes são um tipo de alteração genómica em que dois genes independentes, ou duas porções de genes, ficam justapostas, resultando num gene híbrido7,8
- O aparecimento de proteínas de fusão com material oncogénico pode ter origem em eventos de fusão em que estão envolvidos diferentes pares de genes7
Alterações genómicas no FGFR
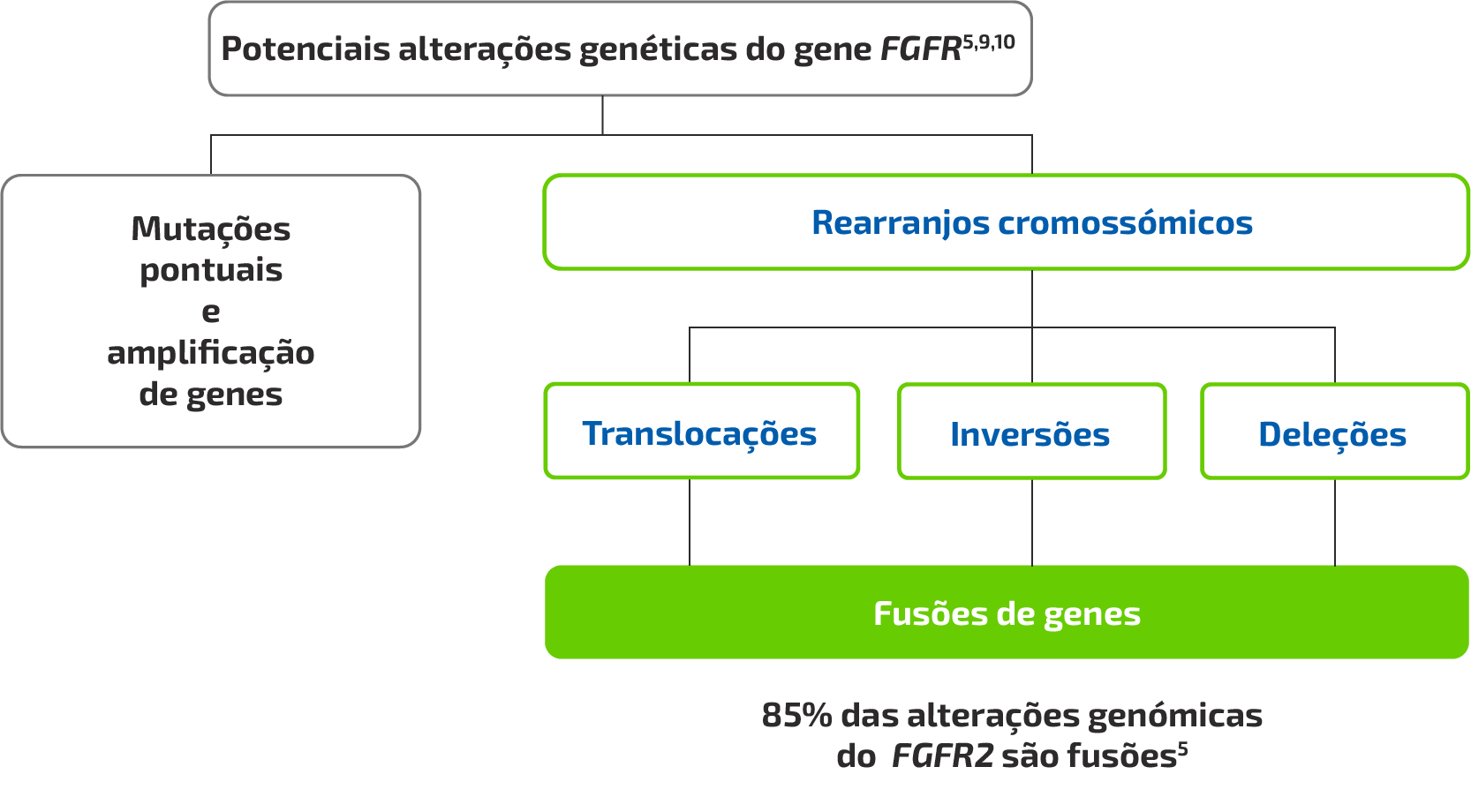
Figura baseada em Jain A, et al. 2018,5 Lowery MA, et al. 2018,9 e Shibata T, et al. 2018.10

 Vantagens
Vantagens
 Desafios
Desafios
