Alterações genómicas no colangiocarcinoma intra-hepático (iCCA)
Foram identificadas alterações genómicas com potenciais implicações terapêuticas em cerca de 50% dos doentes com colangiocarcinoma intra-hepático (iCCA), o que permite traçar um perfil mais individualizado da biologia do tumor de cada doente1
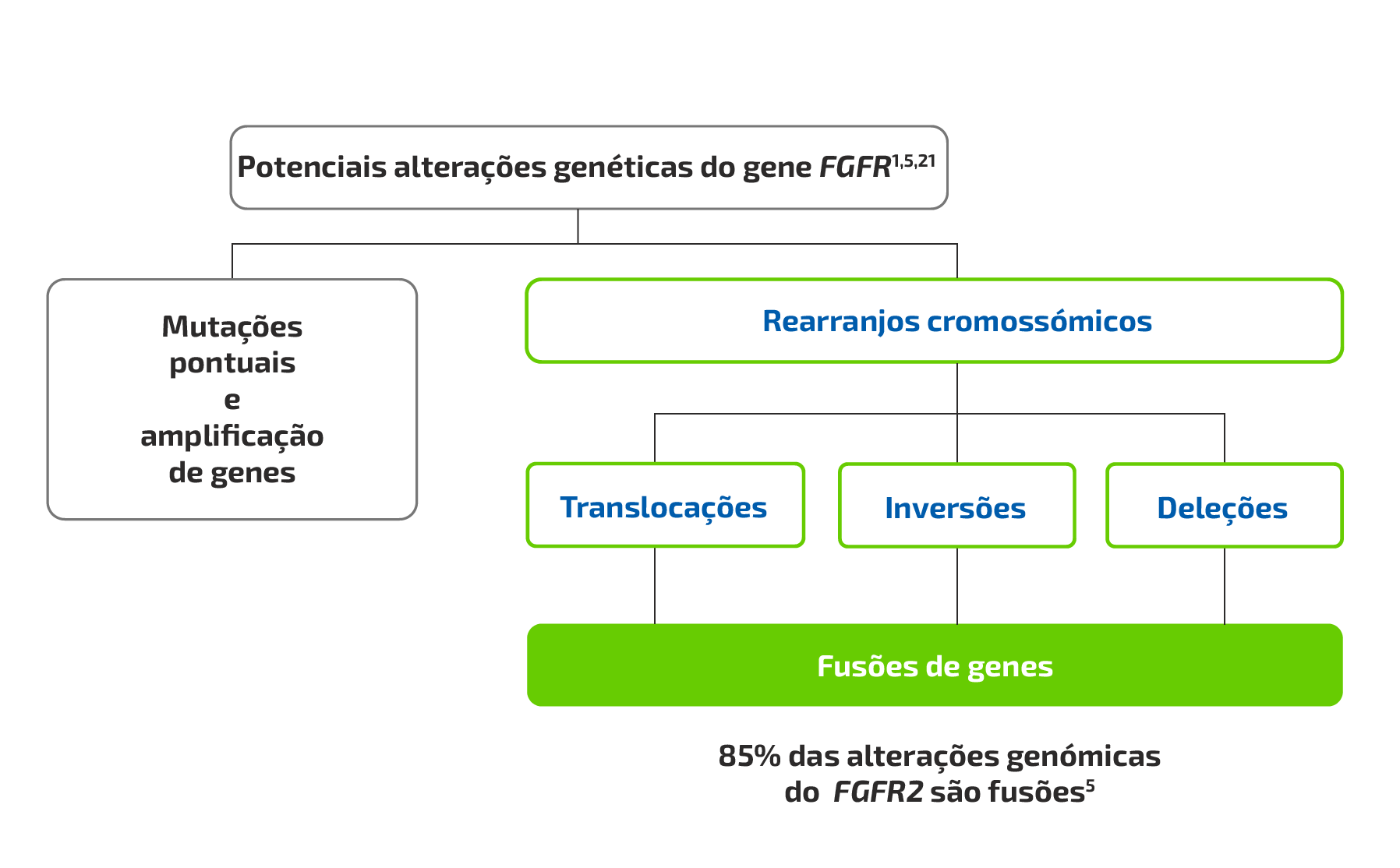
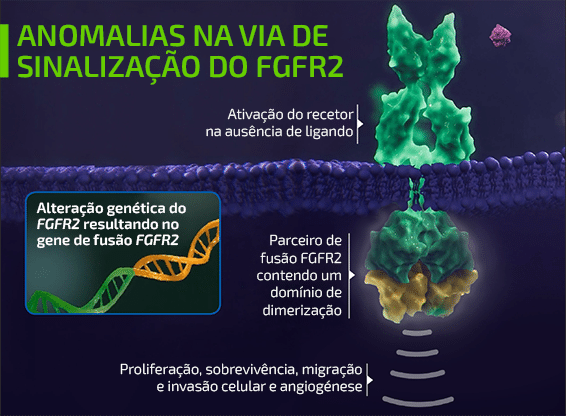
- Duas das alterações genómicas mais frequentes associadas ao iCCA incluem as mutações na isocitrato desidrogenase (IDH) e as fusões do recetor do fator de crescimento de fibroblastos 2 (FGFR2)1,2
São várias as alterações genómicas no iCCA que podem ser usadas como alvos terapêuticos*
*As percentagens apresentadas para cada uma das alterações têm origem em diferentes análises primárias e não provêm de uma única população de doentes com iCCA. Embora as fusões FGFR2 e as mutações IDH tendam a ser mutuamente exclusivas no iCCA, as mutações na categoria “outras alterações” podem ser encontradas em tumores que também apresentam outras mutações ou alterações genéticas que podem ser usadas como alvos terapêuticos.2
BRAF: proto-oncogene B-Raf; EGFR: recetor do fator de crescimento epidérmico; FGFR2: recetor do fator de crescimento de fibroblastos 2; iCCA: colangiocarcinoma intra-hepático; IDH: isocitrato desidrogenase; KRAS: homólogo do oncogene viral do sarcoma de rato Kirsten; MSI-H: instabilidade de microssatélites elevada; NTRK: recetor tirosina quinase neurotrófico.
Figura baseada em dados de Lowery MA, et al. 2018,1 Ross JS, et al. 2014,2 Farshidfar F, et al. 2017,3 Graham RP, et al. 2014,4 Jain A, et al. 2018,5 e Churi CR, et al. 2014.6